목차:
- 데이터 및 모델 준비
- 데이터 전처리(Data Preprocessing)
- 가중치 초기화(Weight Initialization)
- 배치 정규화(Batch Normalization)
- Regularization (L2/L1/Maxnorm/Dropout)
- 손실 함수(Loss functions)
- 요약 (Summary)
데이터 및 모델 준비
앞 장에서 내적(dot product) 및 비선형성(non-linearity)을 연산을 순차적으로 수행하는 뉴런(Neuron) 모델과 이러한 뉴런들의 다층구조(layers)로 구성된 신경망(Neural Networks)에 대해서 소개하였다. 신경망(Neural Networks) 모델은 선형변환(linear mapping) 결과를 비선형성 변환에 적용하는 과정이 연속적으로 발생하게 되고 따라서 선형분류(Linear Classification) 부분에서 소개한 선형변환(linear mapping)을 확장한 새로운 형태의 score function 정의를 필요로 한다. 이번 장에서는 데이터 전처리(data preprocessing), 파라미터 초기화(weight initialization), 손실 함수(loss function)을 소개한다.
데이터 전처리(Data Preprocessing)
데이터 행렬 X에 대해서 일반적으로 아래의 3가지 전처리 방법을 사용한다. (여기서 데이터 X는 D 차원의 데이터 벡터 N개로 이루어진 [N X D] 행렬로 가정한다)
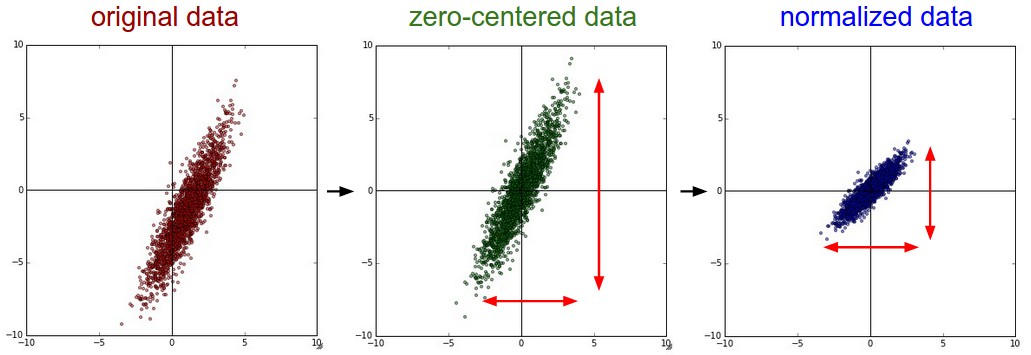
평균 차감(Mean Subtraction)
가장 흔하게 사용되는 데이터 전처리 기법이다. 데이터의 모든 피쳐(feature)에 각각에 대해서 평균값 만큼 차감하는 방법으로 기하학 관점에서 보자면 데이터 군집을 모든 차원에 대해서 원점으로 이동시키는 것으로 해석할 수 있다. numpy에서는 다음과 같이 구현 가능하다: X -= np.mean(X, axis = 0). 특히 이미지 처리에 있어서 계산의 간결성을 위해서 모든 픽셀에서 동일한 값을 차감하는 방식으로 구현한다.(예를들어 numpy에서 X -= np.mean(X))
정규화(Normalization)
정규화(Normalization)는 각 차원의 데이터가 동일한 범위내의 값을 갖도록 하는 전처리 기법을 의미한다. 일반적으로 다음의 2가지 중 하나를 선택하여 구현한다. (1) 각 데이터값을 평균 만큼 차감 하고 표준편차 값으로 나눈다: (X /= np.std(X, axis = 0)), 이때 각 차원에 대해서 개별적으로 연산을 수행한다. (2) 또 다른 기법은 각 차원에서 최소/최대 값이 각각 -1/1의 값을 갖도록 정규화 하는 것이다. 하지만 이 기법은 스케일(scale)(혹은 단위(units))이 다른 features가 (거의) 동일한 비중으로 학습 결과에 영향을 줄 것이라는 가정하에 사용하는 것이 일반적이다.
이미지 처리에서는 각 픽셀 값이 이미 동일한 스케일(0~255)을 갖고 있는 경우가 대부분 이기 때문에 정규화 전처리 기법을 반드시 사용해야 하는 것은 아니다.
PCA와 Whitening 먼저 평균차감(Mean Subtraction) 기법을 이용하여 데이터를 정규화 시킨다. 그리고 데이터 간의 상관관계를 나타내는 공분산(Covariance)을 계산한다:
# Assume input data matrix X of size [N x D]
X -= np.mean(X, axis = 0) # zero-center the data (important)
cov = np.dot(X.T, X) / X.shape[0] # get the data covariance matrix
공분산(Convairance) 행렬에서 (i, j) 값은 X 행렬에서 i번째, j번째 데이터 간의 상관정도(covariance)를 나타내는 값이라고 해석할 수 있다. 특히, 공분산(Covariance) 행렬에서 대각선 상(the diagonal)의 값들은 X 행렬의 각 데이터(주, row 벡터)의 분산(variance)값과 같다. 또한 공분산(Covariance) 행렬은 simmetric, positive semi-definite 성질을 갖는다. 공분산(Covariance) 행렬의 SVD factorication은 다음과 같이 구할 수 있는데,
U,S,V = np.linalg.svd(cov)
여기서 U 행렬의 컬럼(column) 벡터는 아이겐벡터(eigenvector), S는 특이값(singular value)의 1차원 배열이다 (공분산(Covariance)은 symmetric, positive semi-definite의 성질이 있으므로 S 벡터의 각 성분은 아이겐밸류(engienvalue) 제곱의 값을 갖는다) 데이터 X를 고유기저(eigenbasis)에 사상시킴으로써 데이터 간의 상관관계를 없앨 수 있다:
Xrot = np.dot(X, U) # decorrelate the data
U 행렬의 컬럼 벡터는 norm 값은 1이고 서로 직교하는 정규직교(orthonormal)의 성질을 갖고 있기때문에, 기저벡터(basis vector)가 됨을 알 수 있다. 따라서 고유기저(eigenbasis)로 사상(projection)하는 것은 아이겐벡터(eigenvector)를 새로운 축으로하여 X 데이터를 회전하는 것으로 해석할 수 있다. (위의 python 코드에서) Xrot 행렬의 공분산(Covariance)을 구하면 대각행렬(diagonal matrix)인 것을 알 수 있디. np.linalg.svd의 이점 중 하나는 U 행렬의 컬럼 벡터는 각 벡터에 상응하는 아이겐밸류(eigenvalue)의 내림차순으로 정렬된 다는 것이다. 따라서 처음 몇 개의 벡터만 사용하여 데이터 차원을 축소하는데 사용할 수 있다.(and discarding the dimensions along which the data has no variance) 이러한 기법을 Principal Component Analysis (PCA) 차원 축소 기법이라 부르기도 한다.
Xrot_reduced = np.dot(X, U[:,:100]) # Xrot_reduced becomes [N x 100]
위의 연산을 통하여, [N x D] 크기의 X 데이터를 [N x 100] 크기의 데이터로 압축 할 수 있는데 데이터의 variance가 가능한 큰 값을 갖도록 하는 100개의 차원이 선택된다. PCA-축소 기법으로 전처리 된 데이터를 선형 분류기 혹은 신경망에 학습시킴으로써 좋은 성능을 기대할 수 있을 뿐만 아니라 트레이닝 시간과 사용 메모리 용량에서도 이득을 볼 수 있다.
마지막으로 살펴볼 기법은 화이트닝(whitening)으로 이는 기저벡터(eigenbasis) 데이터를 아이겐밸류(eigenvalue) 값으로 나누어 정규화는 기법이다. 화이트닝 변환의 기하학적 해석은 만약 입력 데이터가 multivariable gaussian 분포를라면 화이트닝된 데이터는 평균은 0이고 공분산(covariance)는 단위행렬을 갖는 정규분포를 갖게된다. 와이트닝은 다음과 같이 구할 수 있다:
# whiten the data:
# divide by the eigenvalues (which are square roots of the singular values)
Xwhite = Xrot / np.sqrt(S + 1e-5)
주의: 노이즈 과장(Exaggeratin noice) 위의 식에서 분모가 0이 되는 것을 방지하기 위해서 1e-5(또는 임의의 작은 상수도 무방)를 더한 것에 주목하자. 화이트닝 기법의 단점 중 하나는 모든 차원의 데이터를 동일하게 늘리게 되는데 특히 분산값이 매우 작아 노이즈로 해석할 수 있는 차원의 데이터까지 포함되어 데이터 내의 노이즈 과장되는 효과가 나타난다는 것이다. 이런 경우 보통 (1e-5와 같은 작은 수가 아닌) 큰 수를 분모에 더하는 방식으로 스무딩(smoothing) 효과를 추가하여 이러한 노이즈 과장 현상을 완화 할 수 있다.
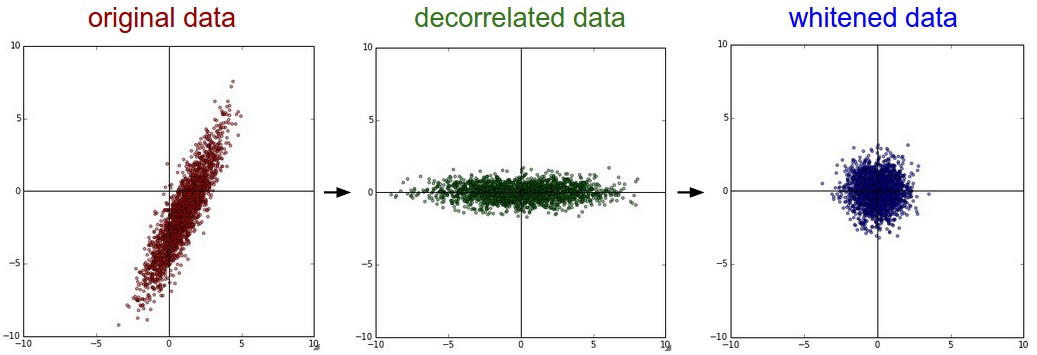
CIFAR-10 이미지에 위에서 소개된 변환들을 적용하여 각 변환 효과를 시각화 할 수 있다. CIFAR-10 학습 데이터는 50,000 x 3072 크기이며 각 이미지 데이터는 3072 차원을 갖는 row 벡터로 표현되어 있다. [3072 x 3072] 크기를 갖는 공분산(covariance) 행렬을 구하고 SVD 분해 (연산 시간이 비교적 오래걸린다)를 한다. 연산을 통하여 구해진 eigenvector는 어떤 특성을 보이는가? 다음의 이미지를 통하여 그 결과를 확인해 볼 수 있다:

실전 응용 모든 변환 기법을 소개하기 위해 PCA/화이트닝(Whitening)도 함께 살펴보았지만 콘볼루션 신경망(Convolutional Networks)에서는 이 변환을 사용하는 경우는 거의 없다. 하지만 (평균차감(Mean Subtraction) 기법을 통하여) zero-centered 데이터로 변환하거나 각 픽셀 값을 정규화 하는 기법은 일반적으로 흔하게 쓰는 전처리 기법 중에 하나이다.
흔히 하는 실수. 전처리 기법을 적용함에 있어서 명심해야 하는 중요한 사항은 전처리를 위한 여러 통계치들은 학습 데이터만 대상으로 추출하고 검증, 테스트 데이터에 적용해야 한다. 예를들어 평균차감(mena subtraction) 기법을 적용 할 때 흔히 하는 실수 중에 하나는 전체 데이터를 대상으로 평균차감 처리를 하고 이 데이터를 학습, 검증, 테스트 데이터로 나누어 사용하는 것이다. 올바른 방법은 학습, 검증, 테스트를 위한 데이터를 먼저 나눈 후에 학습 데이터를 대상으로 평균값을 구한 후에 평균차감 전처리를 모든 데이터군(학습, 검증, 테스트)에 적용하는 것이다.
가중치 초기화
우리는 지금까지 신경망(Neural Network) 구조 및 데이터 전처리 기법에 대해 알아 보았다. 실제 데이터를 신경망 내에서 학습 시키기 전에 해야하는 작업이 있는데 바로 파라미터(paramters) 초기화 이다.
실수: 0으로 초기화하기. 실은 우리가 하지 말아야 하는 방식을 먼저 적용해보자. 학습된 신경망에서 가중치들이 최종적으로 어떤 값으로 수렴해야 하는지 알 수 없지만 데이터 정규화 기법을 적절하게 적용하여 가중치의 절반은 양수 값 나머지 절반은 음수 값을 갖는다는 가정을 할 수 있을 것이다. 더나아가 모든 가중치를 0으로 초기화 함으로써 최상의 학습 결과를 얻을 것이라는 아이디어 또한 합리적인 추론으로 보일 수 있다. 하지만 이러한 방법은 명백히 잘못된 방법이라는 것이 밝혀졌다. 왜냐하면 가중치가 0으로 초기화된 신경망 내의 뉴런들은 모두 동일한 연산 결과를 낼 것이고 따라서 backpropagaton 과정에서 동일한 그라디언트(gradient) 값을 얻게 될 것이고 결과적으로 모든 파라미터(paramter)는 동일한 값으로 업데이트 될 것이기 때문이다. 다시말해, 모든 가중치 값이 동일한 값으로 초기화 된다면 뉴런들의 비대칭성(asymmetry)를 야기할 요소가 사라지게 된다.
0에 가까운 작은 난수. 위에서 언급한 이야기를 종합하자면, 가중치 값은 가능한 0에 가까운 값이어야 또한 모든 동일하게 0이되어서는 안된다는 것이다. 소위 symmetry breaking을 사용하는데 이는 0에 가까운 (하지만 0이 아닌) 값으로 가중치를 초기화시키는 방법이다. 즉, 모든 가중치들을 난수를 이용하여 고유한 값으로 초기화 함으로써 각 파라미터 값이 서로 다른 값으로 업데이트 되고 결과적으로 전체 신경망 내에서 서로 다른 특성을 보이는 다양한 부분으로 분화될 수 있다. 가중치 배열은 다음과 같이 구현할 수 있는데 W = 0.01* np.random.randn(D,H) 여기서 randn은 평균 0, 표준편자 1인 정규 분포로 부터 얻는 값이다. 앞의 공식에 의한면, 모든 가중치 벡터는 다차원 정규 분포로 부터 추출된 벡터로 초기화 되기 때문에 공간 상에서 각 벡터들은 (특정한 패턴 혹은 방향성 없이) 무작위의 방향성을 갖게 된다. 정규 분포가 아닌 균일 분포(uniform distribution)로 부터 추출된 값으로 가중치를 초기화 해도 무방하지만 이 방법은 학습된 최종 성능에 미치는 영향은 미미한 것으로 알려져 있다.
주의: 가중치를 0에 가까운 작은 값으로 초기화 하는 것은 항상 좋은 성능을 답보하는 것은 아니다. 예를들어 아주 작은 값으로 구성된 가중치 값으로 된 신경망의 경우 backpropagation 연상 과정에서 그라디언트(gradient) 또한 작은 값을 갖게 된다(그라디언트(gradient)는 가중치 값에 례하기 때문). 이는 네트워크의 역방향으로 흐르며 전달되는 “그라디언트 시그널(gradient signal)”을 감소시키게 되고 이는 신경망 학습에 있어서 중요한 문제를 야기하게 된다.
분산 보정, 1/sqrt(n). 위에서 제안한 방법의 문제점 중 하나는 랜덤값으로 초기화된 뉴런으로 학습되어 나온 결과의 분포가 입력 데이터 수에 비례하여 커지는 분산을 갖는다는 것이다. 가중치 벡터를 팬인(fan-in)(입력 데이터 수)의 제곱근 값으로 나누는 연산을 통하여 뉴런 출력의 분산이 1로 정규화 할 수 있다. 권장되는 휴리스틱(heuristic) 기법은 뉴런의 가중치 벡터를 다음과 같이 초기화 하는 것이다. w = np.random.randn(n) / sqrt(n) (n: 입력 수). 이 방법은 근사적으로 동일한 출력 분포를 갖게 할 뿐만 아니라 신경망의 수렴률 또한 향상시키는 것으로 알려져 있다.
이는 다음의 유도 과정을 통해서 확인할 수 있다.: 가중치 값을 나타내는 와 입력 데이터를 나타내는 의 내적 연산 가 있다고 하자. 이는 비선형 연산 이전 단계에 일어나는 뉴런 연산이 되고 의 연산은 다음과 같이 구할 수 있다.
처음 2단계는 분산의 성질을 이용하여 전개하였다. 가중치와 입력 데이터 모두 평균이 0이라고 가정하고 있기때문에 이 되고 따라서 3번째 단계에서 4번째 단계로 전개가 가능하다. 하지만 평균이 0이라고 가정하는 것은 일반적으로 모든 상황에서 가정할 수 있는 것은 아니라는 것을 명심해야 한다. 일례로 ReLU 유닛은 0보다 큰 평균값을 갖는다. 마지막 단계는 모두 동일한 확률 분포(identically distribution)를 갖는다고 가정하여 전개할 수 있다.
위의 유도 과정을 통하여 가 입력 데이터 와 동일한 분산을 갖기 위해서는 초기화 단계에서 모든 가중치 벡터 의 분산이 로 만들어야 한다는 것을 알 수 있다. 또한 확률 변수 , 스칼라(scalar) 값 에 대해서 이 성립하므로 분산이 이 되기 위해서는 표준정규분포에서 값을 뽑아서 곱해주어야 한다는 것을 알 수 있다. w = np.random.randn(n) / sqrt(n)로 가중치를 초기화하면 된다.
이와 유사한 내용의 연구를 Understanding the difficulty of training deep feedforward neural networks by Glorot et al. 논문에서 확인할 수 있다. 논문의 저자는 ( 각각 이전 레이어, 다음 레이어의 입력 유닛수)로 초기화 할 것을 권고하며 끝맺고 있다. This is motivated by based on a compromise and an equivalent analysis of the backpropagated gradients. 동일한 주제에 대한 더 최근의 연구는 Delving Deep into Rectifiers: Surpassing Human-Level Performance on ImageNet Classification by He et al.에서 확인 할 수 있는데, 특히 ReLU 뉴런에 대한 초기화 방법에 대해서 다루고 있다. 이 논문에서는 신경망에서 뉴런의 분산은 가 되야 한다고 결론내리고 있다. 즉, w = np.random.randn(n) * sqrt(2.0/n)을 이용하여 가중치를 초기화 하는 것을 의미하며 이는 특히 ReLU 뉴런이 사용되는 신경망에서 최근에 권장되고 있는 방식이다.
희소 초기화(Sparse initialization). 보정되지 않은 분산을 위한 또 다른 방법은 모든 가중치 행렬을 0으로 초기화 하는 것이다. 이때 대칭성을 깨기 위해서 모든 뉴런을 고정된 숫자의 아래 단계 뉴런들과 무작위로 연결한다.(with weights sampled from a small gaussian as above) 연결하는 뉴런의 수는 대략 10개 정도이다.
bias 초기화. 가중치에 랜덤한 값을 설정하므로써 대칭성 문제는 해결되기 때문에 주로bias는 0으로 초기화한다. ReLU 연산의 비선형성에 의해서 몇몇 경우에는 0.01과 같은 작은 상수값을 사용하기도 하는데 이는 ReLU 연산이 초기부터 fire되고 따라서 그라디언트(gradient) 값이 유미의한 값을 갖고 신경망을 통해서 전달되는 것을 보장할 수 있기 때문이다. 하지만 상수값을 사용하는 방식이 성능 향상을 언제나 보장하는 것인가에 대해서는 이견이 존재한다(실제 몇몇 사례에서 더 나쁜 결과가 볼 수 있다). 따라서 bias 값은 0으로 초기화 하는 것이 더 일반적이라 할 수 있다.
실전응용, ReLU 유닛을 사용하고 w = np.random.randn(n) * sqrt(2.0/n) 초기화하는 것이 요즘의 추세이다 He et al..
배치 정규화(Batch Normalization) 최근 Ioffe and Szegedy에 의해서 제안된 배치 정규화(Batch Normalization) 기법은 신경망 학습단계에서 activation 값이 표준정규분포를 갖도록 강제하는 기법으로 신경망을 적절한 값으로 초기화하여 그동안 많은 연구자들을 괴롭혀왔던 초기화 문제의 상당부분을 해소해 주었다. 여기서 사용한 정규화 기법이 단순 미분 가능한 연산이었기에 적용 가능하다. 실제 구현에서는 배치 정규화 레이어를 fully-connected 레이어 (혹은 곧 설명하게될 컨볼루션 레이어) 다음, 비선형 연산 이전에 위치 시키는 방식으로 이 기법을 신경망에 적용할 수 있다. 앞에서 링크된 논문에서 배치 정규화(Batch Normalization) 기법에 대해서 자세하게 설명하고 있기 때문에 여기에서는 관련 기법을 자세하게 다루지는 않겠지만, 이 기법은 이미 신경망 학습에서 일반적으로 사용되는 기법중 하나라는 것을 밝혀두는 바이다. 실제 적용 사례를 보면 배치 정규화(Batch Normalization)을 사용하여 학습한 신경망은 특히 나쁜 초기화의 영향에 강하다는 것이 밝혀졌다. 배치 정규화(Batch Normalization)는 신경망 내의 모든 레이어에서 전처리 과정을 수행하는 것이지만, 미분 가능하다는 성질에 의해서 신경망 내의 학습 단계로 통합되었다고 볼 수 있다.
Regularization
이번 파트에서는 신경망 학습에서 overfitting을 막을 수 있는 몇가지 방법을 소개하고자 한다.
L2 regularization은 가장 일반적으로 사용되는 regularization 기법이다. 모든 파라미터 제곱 만큼의 크기를 목적 함수에 제약을 거는 방식으로 구현된다. 다시말해, 가중치 벡터 가 있을때, 목적 함수에 를 더한다 (여가서 는 regulrization의 강도를 의미). 부분이 항상 존재하는데 이는 앞서 본 regularization 값을 로 미분했을 때 가 아닌 의 값을 갖도록 하기 위함이다. L2 reguralization은 큰 값이 많이 존재하는 가중치에 제약을 주고, 가중치 값을 가능한 널리 퍼지도록 하는 효과를 주는 것으로 볼 수 있다. 선형 분류(Linear Classification) 장에서도 이야기 했던 가중치와 입력 데이터가 곱해지는 연산이므로 특정 몇개의 입력 데이터에 강하게 적용되기 보다는 모든 입력데이터에 약하게 적용되도록 하는 것이 일반적이다. gradient descent 업데이트 과정에서 L2 regularization을 적용하는 것은 모든 가중치 값이 선형적으로 감소하게 된다: W += -lambda * W이 0으로 감소하게 된다.
L1 regularization 또한 상대적으로 많이 사용되는 regularization 기법으로 가중치 벡터가 있을때, 목적 함수에 를 더한다. 다음과 같이 L1 regularization과 L2 regularization을 동시에 사용할 수도 있다: (Elastic net regularization라고도 불린다). L1 regularization은 최적화 과정 동안 가중치 벡터들을 sparse하게(거의 0에 가깝게) 만드는 흥미로운 특성이 있다. 다시 말해, L1 regularization이 적용된 뉴런들은 결국 입력 데이터의 sparse한 부분만을 사용하고, “noisy” 입력 데이터에 거의 영향을 받지 않는다. 이에 반해, L2 regularization을 적용하면 최종 가중치 벡터들은 작은 값들이 퍼져있는 형태로 나타나게 된다. 실제 신경망 학습에 적용할 때, 만약 특정한 feature selection 후 학습하는 것이 아니라면 많은 경우에 L2 regularization을 사용하면 훨씬 좋은 성능을 기대할 수 있다.
Max norm constrains. regularizatio 기법 중 하나로 가중치 벡터의 길이가 미리 정해 놓은 상한 값을 넘지 못하도록 제한하면서 gradient descent 연산도 제한 된 조건 안에서만 계산하도록 하는 projected gradient descent를 사용한다. 신경망 학습에 실제 적용하는 방법은, 먼저 일반적인 방법으로 파라미터를 업데이트 하고, 모든 뉴런의 가중치 벡터 이 대해서 를 만족하도록 제한을 가한다. 일반적으로 c값은 3 혹은 4로 설정한다. 이 regularization 기법을 적용한 몇몇 연구를 통하여 성능 향상이 있음이 알려졌다. 이 기법의 흥미로운 사실 중 하나는 학습률(learning rate)을 큰 값을로 설정하고 학습 시키더라도 신경망이 “explode”하지 않는 다는 것인데 이는 업데이트 될 때마다 제한된 범위 내의 값을 갖기 때문이다.
Dropout Dropout: A Simple Way to Prevent Neural Networks from Overfitting에서 Srivastava et al.의해 최근 제안된 기법으로 간단하지만 아주 효과적인 regularization 방법으로 위에서 소개한 다른 regularization 기법들과 (L1, L2, maxnorm) 상호 보완적인 방법으로 알려져 있다. 각 뉴런들을 의 확률로 활성화 시켜 학습에 적용 하는 방식으로 구현할 수 있다.
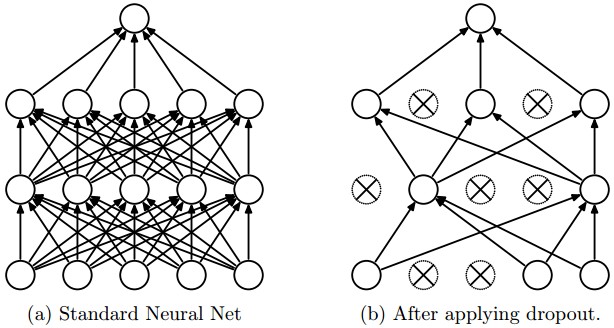
3-레이어 신경망 회로에 적용된 Vanilla dropout 예제를 아래 구현하였다.
""" Vanilla Dropout: Not recommended implementation (see notes below) """
p = 0.5 # probability of keeping a unit active. higher = less dropout
def train_step(X):
""" X contains the data """
# forward pass for example 3-layer neural network
H1 = np.maximum(0, np.dot(W1, X) + b1)
U1 = np.random.rand(*H1.shape) < p # first dropout mask
H1 *= U1 # drop!
H2 = np.maximum(0, np.dot(W2, H1) + b2)
U2 = np.random.rand(*H2.shape) < p # second dropout mask
H2 *= U2 # drop!
out = np.dot(W3, H2) + b3
# backward pass: compute gradients... (not shown)
# perform parameter update... (not shown)
def predict(X):
# ensembled forward pass
H1 = np.maximum(0, np.dot(W1, X) + b1) * p # NOTE: scale the activations
H2 = np.maximum(0, np.dot(W2, H1) + b2) * p # NOTE: scale the activations
out = np.dot(W3, H2) + b3
train_step 함수를 보면 첫번째 히든 레이어와 두번째 히든레이어 총 2 부분에서 dropout이 적용된 것을 볼 수 있다. 물론 입력 데이터 X를 위한 p=0.5 마스크를 만들어 입력 단에도 dropout을 적용할 수 있다. 역전파(backward pass) 과정에서는 forward에서 사용된 U1, U2를 사용하여 수행한다.
predict 함수을 보면 dropout을 적용하지 않았지만 히든 레이어 출력 데이터에 만큼 스케일링 한 것을 주목할 필요가 있다. 테스트 과정에서 모든 뉴런은 모든 입력 데이터를 받기 때문에 학습 과정에서 얻을 수 있는 출력값과 동일한 조건으로 맞추어 보정해야한다. dropout 확률 인 경우를 가정해 보자. 테스트 과정 동안 뉴런의 출력 값은 모두 1/2만큼 줄어들어야 하는데 이는 학습 과정 동안 뉴런 출력 데이터의 기대값과 동일하게 맞추기 위함이다. 뉴런 가 있을때 dropout 적용하지 않은 출력 데이터가 있다고 가정하자. dropout을 적용하면 이 뉴런에서의 기대값은 가 되는데 이는 의 확률로 뉴런의 출력 데이터 값이 0이 되기 때문이다. 테스트 과정에서는 모든 뉴런을 사용하기 때문에 동일한 기대값을 갖기 위해서는 로 보정해 주어야 한다. 또 다른 관점에서 보면 만큼 값을 줄이는 과정은 모든 가능한 dropout 마스크를 적용한 후 그 결과를 이용하여 ensemble prediction을 수행하는 것으로 해석 할 수 있다.
위에서 소개한 방법은 테스트 과정에서 뉴런 출력에 를 곱하는 연산이 수행해야 하는데 이는 원하지 않는 방식인 경우가 많다. 테스트 과정에서의 성능은 매우 중요한 이슈이기 때문에 많은 경우에 inverted droptou 방식이 더 선호된다. 이는 스케일링 연산을 학습 과정에서 적용하고 테스트 과정에서는 추가적인 스케일링 연산없이 바로 사용하는 방식이다. 이 기법의 또 다른 장점은 만약 dropout을 수정하기로 했을때 prediction 코드에는 여전히 변화가 없다는 것이다. Inverted dropout은 다음과 같이 구현할 수 있다.
"""
Inverted Dropout: Recommended implementation example.
We drop and scale at train time and don't do anything at test time.
"""
p = 0.5 # probability of keeping a unit active. higher = less dropout
def train_step(X):
# forward pass for example 3-layer neural network
H1 = np.maximum(0, np.dot(W1, X) + b1)
U1 = (np.random.rand(*H1.shape) < p) / p # first dropout mask. Notice /p!
H1 *= U1 # drop!
H2 = np.maximum(0, np.dot(W2, H1) + b2)
U2 = (np.random.rand(*H2.shape) < p) / p # second dropout mask. Notice /p!
H2 *= U2 # drop!
out = np.dot(W3, H2) + b3
# backward pass: compute gradients... (not shown)
# perform parameter update... (not shown)
def predict(X):
# ensembled forward pass
H1 = np.maximum(0, np.dot(W1, X) + b1) # no scaling necessary
H2 = np.maximum(0, np.dot(W2, H1) + b2)
out = np.dot(W3, H2) + b3
dropout이 처음 소개된 이후로 실제 적용 사례에서 나타난 성능 향상의 근본 원인과 기존의 다른 regularization 기법과의 관계등에 대한 수많은 연구가 진행되었다. 관련하여 다음의 자료들을 읽어보는 것인 도움이 될 것이라 생각된다:
- Dropout 논문 by Srivastava et al. 2014.
- Dropout Training as Adaptive Regularization: “we show that the dropout regularizer is first-order equivalent to an L2 regularizer applied after scaling the features by an estimate of the inverse diagonal Fisher information matrix”.
forward pass에서 노이즈 관련하여 넓은 의미에서 보자면 dropout은 신경망의 forward pass에서 stochastic(확률적) 접근을 도입하는 것으로 볼 수 있다. testing 과정에서 노이즈 감소하게 되는데 이는 분석적 해석은 확률 $p$ 만큼 곱해진 결과라고 볼 수 있고, 수치적 해석은 랜덤하게 선택된 forward pass를 여러차례 수행한 결과의 평균이라고 볼 수 있다. 동일한 관점에서의 연구들 중 하나인 DropConnect를 보면 forward pass 동안 가중치 값을 0으로 설정하는 것으로 볼 수 있다. Convolutional 신경망에서 dropout과 함께 stochastic(확률적) 풀링(pooling), 부분 풀링, 데이터 augmentation 등의 기법을 같이 사용하여 추가적인 성능 향상을 기대할 수 있다. 이에 대해서는 뒤에서 더 자세히 살펴 볼 것이다.
Bias regularization. Linear Classification 파트에서 설명했듯이, bias 텀은 regularization을 적용하지 않는 것이 일반적인데, 이는 학습된 가중치와 곱셈 연산을 하지 않기 때문에 목적 함수에서 데이터 dimension을 결정하는 요소로 작용하지 않는다. 그러나 실제 적용 사례들을 보면 bias 텀에 regularization을 적용하였을 때 심각한 성능 저하가 나타나는 경우는 극히 드문 것으로 알려져 있다. 이는 모든 가중치 텀의 갯수와 비교했을 때 bais 텀의 갯수는 무시할 만한 수준이어서 so the classifier can “afford to” use the biases if it needs them to obtain a better data loss.
레이어별 정규화. 마지막 출력 레이어를 제외하고 레이어를 각각 따로 정규화 하는 것은 일반적인 방법이 아니다. 레이어 별 정규화를 적용한 논문수도 상대적으로 매우 적은 편이다.
실전 응용: 하나의 공통된 L2 정규화를 사용하는 것이 일반적이다. 또한 모든 레이어 이후에 dropout을 적용하는 것 또한 일반적으로 많이 사용된다. dropout rate로 $p = 0.5*이 주로 사용되지만 validation 과정에서 값을 조정하기도 한다.
Loss functions
We have discussed the regularization loss part of the objective, which can be seen as penalizing some measure of complexity of the model. The second part of an objective is the data loss, which in a supervised learning problem measures the compatibility between a prediction (e.g. the class scores in classification) and the ground truth label. The data loss takes the form of an average over the data losses for every individual example. That is, $L = \frac{1}{N} \sum_i L_i$ where $N$ is the number of training data. Lets abbreviate $f = f(x_i; W)$ to be the activations of the output layer in a Neural Network. There are several types of problems you might want to solve in practice:
Classification is the case that we have so far discussed at length. Here, we assume a dataset of examples and a single correct label (out of a fixed set) for each example. One of two most commonly seen cost functions in this setting are the SVM (e.g. the Weston Watkins formulation):
As we briefly alluded to, some people report better performance with the squared hinge loss (i.e. instead using $\max(0, f_j - f_{y_i} + 1)^2$). The second common choice is the Softmax classifier that uses the cross-entropy loss:
Problem: Large number of classes. When the set of labels is very large (e.g. words in English dictionary, or ImageNet which contains 22,000 categories), it may be helpful to use Hierarchical Softmax (see one explanation here (pdf)). The hierarchical softmax decomposes labels into a tree. Each label is then represented as a path along the tree, and a Softmax classifier is trained at every node of the tree to disambiguate between the left and right branch. The structure of the tree strongly impacts the performance and is generally problem-dependent.
Attribute classification. Both losses above assume that there is a single correct answer $y_i$. But what if $y_i$ is a binary vector where every example may or may not have a certain attribute, and where the attributes are not exclusive? For example, images on Instagram can be thought of as labeled with a certain subset of hashtags from a large set of all hashtags, and an image may contain multiple. A sensible approach in this case is to build a binary classifier for every single attribute independently. For example, a binary classifier for each category independently would take the form:
where the sum is over all categories $j$, and $y_{ij}$ is either +1 or -1 depending on whether the i-th example is labeled with the j-th attribute, and the score vector $f_j$ will be positive when the class is predicted to be present and negative otherwise. Notice that loss is accumulated if a positive example has score less than +1, or when a negative example has score greater than -1.
An alternative to this loss would be to train a logistic regression classifier for every attribute independently. A binary logistic regression classifier has only two classes (0,1), and calculates the probability of class 1 as:
Since the probabilities of class 1 and 0 sum to one, the probability for class 0 is $P(y = 0 \mid x; w, b) = 1 - P(y = 1 \mid x; w,b)$. Hence, an example is classified as a positive example (y = 1) if $\sigma (w^Tx + b) > 0.5$, or equivalently if the score $w^Tx +b > 0$. The loss function then maximizes the log likelihood of this probability. You can convince yourself that this simplifies to:
where the labels $y_{ij}$ are assumed to be either 1 (positive) or 0 (negative), and $\sigma(\cdot)$ is the sigmoid function. The expression above can look scary but the gradient on $f$ is in fact extremely simple and intuitive: $\partial{L_i} / \partial{f_j} = y_{ij} - \sigma(f_j)$ (as you can double check yourself by taking the derivatives).
Regression is the task of predicting real-valued quantities, such as the price of houses or the length of something in an image. For this task, it is common to compute the loss between the predicted quantity and the true answer and then measure the L2 squared norm, or L1 norm of the difference. The L2 norm squared would compute the loss for a single example of the form:
The reason the L2 norm is squared in the objective is that the gradient becomes much simpler, without changing the optimal parameters since squaring is a monotonic operation. The L1 norm would be formulated by summing the absolute value along each dimension:
where the sum $\sum_j$ is a sum over all dimensions of the desired prediction, if there is more than one quantity being predicted. Looking at only the j-th dimension of the i-th example and denoting the difference between the true and the predicted value by $\delta_{ij}$, the gradient for this dimension (i.e. $\partial{L_i} / \partial{f_j}$) is easily derived to be either $\delta_{ij}$ with the L2 norm, or $sign(\delta_{ij})$. That is, the gradient on the score will either be directly proportional to the difference in the error, or it will be fixed and only inherit the sign of the difference.
Word of caution: It is important to note that the L2 loss is much harder to optimize than a more stable loss such as Softmax. Intuitively, it requires a very fragile and specific property from the network to output exactly one correct value for each input (and its augmentations). Notice that this is not the case with Softmax, where the precise value of each score is less important: It only matters that their magnitudes are appropriate. Additionally, the L2 loss is less robust because outliers can introduce huge gradients. When faced with a regression problem, first consider if it is absolutely inadequate to quantize the output into bins. For example, if you are predicting star rating for a product, it might work much better to use 5 independent classifiers for ratings of 1-5 stars instead of a regression loss. Classification has the additional benefit that it can give you a distribution over the regression outputs, not just a single output with no indication of its confidence. If you’re certain that classification is not appropriate, use the L2 but be careful: For example, the L2 is more fragile and applying dropout in the network (especially in the layer right before the L2 loss) is not a great idea.
When faced with a regression task, first consider if it is absolutely necessary. Instead, have a strong preference to discretizing your outputs to bins and perform classification over them whenever possible.
Structured prediction. The structured loss refers to a case where the labels can be arbitrary structures such as graphs, trees, or other complex objects. Usually it is also assumed that the space of structures is very large and not easily enumerable. The basic idea behind the structured SVM loss is to demand a margin between the correct structure $y_i$ and the highest-scoring incorrect structure. It is not common to solve this problem as a simple unconstrained optimization problem with gradient descent. Instead, special solvers are usually devised so that the specific simplifying assumptions of the structure space can be taken advantage of. We mention the problem briefly but consider the specifics to be outside of the scope of the class.
Summary
In summary:
- The recommended preprocessing is to center the data to have mean of zero, and normalize its scale to [-1, 1] along each feature
- Initialize the weights by drawing them from a gaussian distribution with standard deviation of $\sqrt{2/n}$, where $n$ is the number of inputs to the neuron. E.g. in numpy:
w = np.random.randn(n) * sqrt(2.0/n). - Use L2 regularization and dropout (the inverted version)
- Use batch normalization
- We discussed different tasks you might want to perform in practice, and the most common loss functions for each task
We’ve now preprocessed the data and set up and initialized the model. In the next section we will look at the learning process and its dynamics.
번역: 서종한 (salopge)